Условно окрасьте точки данных за пределами полос уверенности в R
Я должен окрасить точки данных, которые являются за пределами полосы уверенности на графике ниже по-другому по сравнению с теми, которые в полосах. Я должен добавить отдельный столбец к своему набору данных, чтобы записать, являются ли точки данных в полосах уверенности? Можно ли обеспечить пример?
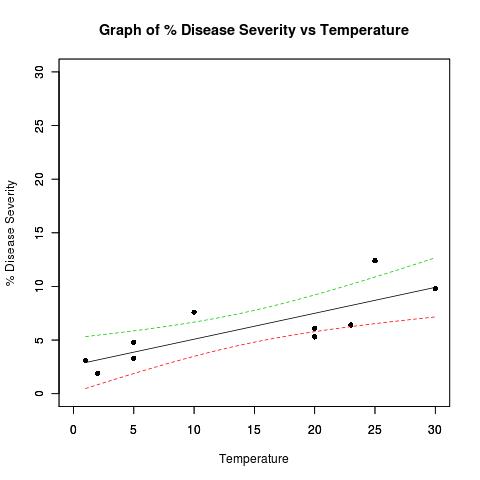
Набор данных в качестве примера:
## Dataset from http://www.apsnet.org/education/advancedplantpath/topics/RModules/doc1/04_Linear_regression.html
## Disease severity as a function of temperature
# Response variable, disease severity
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
# Take a look at the data
plot(
diseasesev~temperature,
data=severity,
xlab="Temperature",
ylab="% Disease Severity",
pch=16,
pty="s",
xlim=c(0,30),
ylim=c(0,30)
)
title(main="Graph of % Disease Severity vs Temperature")
par(new=TRUE) # don't start a new plot
## Get datapoints predicted by best fit line and confidence bands
## at every 0.01 interval
xRange=data.frame(temperature=seq(min(temperature),max(temperature),0.01))
pred4plot <- predict(
lm(diseasesev~temperature),
xRange,
level=0.95,
interval="confidence"
)
## Plot lines derrived from best fit line and confidence band datapoints
matplot(
xRange,
pred4plot,
lty=c(1,2,2), #vector of line types and widths
type="l", #type of plot for each column of y
xlim=c(0,30),
ylim=c(0,30),
xlab="",
ylab=""
)
3 ответа
Вероятно, самый простой способ - вычислить вектор из ИСТИНА / ЛОЖЬ значений, которые указывают, находится ли точка данных внутри доверительного интервала. или не. Я собираюсь немного перетасовать ваш пример, чтобы все вычисления были завершены до выполнения команд построения - это обеспечивает четкое разделение логики программы, которое можно было бы использовать, если бы вы упаковали часть этого в функцию. .
Первая часть почти такая же, за исключением того, что я заменил дополнительный вызов lm () внутри pred () переменной severity.lm . - нет необходимости использовать дополнительные вычислительные ресурсы для пересчета линейной модели, когда она уже хранится:
## Dataset from
# apsnet.org/education/advancedplantpath/topics/
# RModules/doc1/04_Linear_regression.html
## Disease severity as a function of temperature
# Response variable, disease severity
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
## Get datapoints predicted by best fit line and confidence bands
## at every 0.01 interval
xRange=data.frame(temperature=seq(min(temperature),max(temperature),0.01))
pred4plot <- predict(
severity.lm,
xRange,
level=0.95,
interval="confidence"
)
Теперь мы вычислим доверительные интервалы для исходных точек данных и запустим тест, чтобы увидеть, находятся ли точки внутри интервал:
modelConfInt <- predict(
severity.lm,
level = 0.95,
interval = "confidence"
)
insideInterval <- modelConfInt[,'lwr'] < severity[['diseasesev']] &
severity[['diseasesev']] < modelConfInt[,'upr']
Затем мы построим график - сначала функцию высокоуровневого построения plot () , как вы использовали его в своем примере, но мы будем строить только точки внутри интервала .Затем мы продолжим работу с функцией низкого уровня points () , которая будет отображать все точки вне интервала другим цветом. Наконец, matplot () будет использоваться для заполнения доверительных интервалов, как вы его использовали. Однако вместо вызова par (new = TRUE) я предпочитаю передавать аргумент add = TRUE высокоуровневым функциям, чтобы они действовали как низкоуровневые функции.
Использование par (new = TRUE) похоже на подтасовку функции построения графика, которая может иметь непредвиденные последствия. Аргумент add предоставляется многими функциями, чтобы заставить их добавлять информацию к графику, а не перерисовывать его. Я бы рекомендовал использовать этот аргумент, когда это возможно, и прибегать к манипуляциям с par () в крайнем случае.
# Take a look at the data- those points inside the interval
plot(
diseasesev~temperature,
data=severity[ insideInterval,],
xlab="Temperature",
ylab="% Disease Severity",
pch=16,
pty="s",
xlim=c(0,30),
ylim=c(0,30)
)
title(main="Graph of % Disease Severity vs Temperature")
# Add points outside the interval, color differently
points(
diseasesev~temperature,
pch = 16,
col = 'red',
data = severity[ !insideInterval,]
)
# Add regression line and confidence intervals
matplot(
xRange,
pred4plot,
lty=c(1,2,2), #vector of line types and widths
type="l", #type of plot for each column of y
add = TRUE
)
Ну, я думал, что с ggplot2 это будет довольно просто, но теперь я понимаю, что понятия не имею, как рассчитываются пределы достоверности для stat_smooth / geom_smooth.
Примите во внимание следующее:
library(ggplot2)
pred <- as.data.frame(predict(severity.lm,level=0.95,interval="confidence"))
dat <- data.frame(diseasesev,temperature,
in_interval = diseasesev <=pred$upr & diseasesev >=pred$lwr ,pred)
ggplot(dat,aes(y=diseasesev,x=temperature)) +
stat_smooth(method='lm') + geom_point(aes(colour=in_interval)) +
geom_line(aes(y=lwr),colour=I('red')) + geom_line(aes(y=upr),colour=I('red'))
Это дает: альтернативный текст http://ifellows.ucsd.edu/pmwiki/uploads/Main/strangeplot.jpg
Я не понимаю почему доверительный интервал, рассчитанный с помощью stat_smooth, несовместим с диапазоном, рассчитанным непосредственно из прогноза (т. е. красные линии). Может ли кто-нибудь пролить свет на это?
Edit:
понял это. ggplot2 использует стандартную ошибку 1,96 * для рисования интервалов для всех методов сглаживания.
pred <- as.data.frame(predict(severity.lm,se.fit=TRUE,
level=0.95,interval="confidence"))
dat <- data.frame(diseasesev,temperature,
in_interval = diseasesev <=pred$fit.upr & diseasesev >=pred$fit.lwr ,pred)
ggplot(dat,aes(y=diseasesev,x=temperature)) +
stat_smooth(method='lm') +
geom_point(aes(colour=in_interval)) +
geom_line(aes(y=fit.lwr),colour=I('red')) +
geom_line(aes(y=fit.upr),colour=I('red')) +
geom_line(aes(y=fit.fit-1.96*se.fit),colour=I('green')) +
geom_line(aes(y=fit.fit+1.96*se.fit),colour=I('green'))
Мне понравилась идея, и я попытался создать для этого функцию. Конечно, это далеко не идеально. Ваши комментарии приветствуются.
diseasesev<-c(1.9,3.1,3.3,4.8,5.3,6.1,6.4,7.6,9.8,12.4)
# Predictor variable, (Centigrade)
temperature<-c(2,1,5,5,20,20,23,10,30,25)
## For convenience, the data may be formatted into a dataframe
severity <- as.data.frame(cbind(diseasesev,temperature))
## Fit a linear model for the data and summarize the output from function lm()
severity.lm <- lm(diseasesev~temperature,data=severity)
# Function to plot the linear regression and overlay the confidence intervals
ci.lines<-function(model,conf= .95 ,interval = "confidence"){
x <- model[[12]][[2]]
y <- model[[12]][[1]]
xm<-mean(x)
n<-length(x)
ssx<- sum((x - mean(x))^2)
s.t<- qt(1-(1-conf)/2,(n-2))
xv<-seq(min(x),max(x),(max(x) - min(x))/100)
yv<- coef(model)[1]+coef(model)[2]*xv
se <- switch(interval,
confidence = summary(model)[[6]] * sqrt(1/n+(xv-xm)^2/ssx),
prediction = summary(model)[[6]] * sqrt(1+1/n+(xv-xm)^2/ssx)
)
# summary(model)[[6]] = 'sigma'
ci<-s.t*se
uyv<-yv+ci
lyv<-yv-ci
limits1 <- min(c(x,y))
limits2 <- max(c(x,y))
predictions <- predict(model, level = conf, interval = interval)
insideCI <- predictions[,'lwr'] < y & y < predictions[,'upr']
x_name <- rownames(attr(model[[11]],"factors"))[2]
y_name <- rownames(attr(model[[11]],"factors"))[1]
plot(x[insideCI],y[insideCI],
pch=16,pty="s",xlim=c(limits1,limits2),ylim=c(limits1,limits2),
xlab=x_name,
ylab=y_name,
main=paste("Graph of ", y_name, " vs ", x_name,sep=""))
abline(model)
points(x[!insideCI],y[!insideCI], pch = 16, col = 'red')
lines(xv,uyv,lty=2,col=3)
lines(xv,lyv,lty=2,col=3)
}
Используйте это так:
ci.lines(severity.lm, conf= .95 , interval = "confidence")
ci.lines(severity.lm, conf= .85 , interval = "prediction")
